journal of cheminformatics | 基于对比学习的酶促反应分类ai模型
ec分类编号(ec number)是一套对酶催化功能进行系统分类的体系。ec分类编号不仅可以用来描述酶的功能,也可以对相应的酶促反应进行分类。虽然有经验的化学家可以手动分析一个酶促反应并对其ec分类编号的归类,但当面临庞大的待预测数据时,例如逆合成路径规划,药物代谢路径预测等,化学家们也会显得力不从心。此时,一个可靠的自动分类预测模型显得格外重要。
构建酶促反应ec分类编号预测模型面临两个主要瓶颈——酶促反应数据少,数据分布极度不平衡。为了克服这些难题,由中国科学院深圳先进技术研究院的罗小舟领衔的研究团队,近日在journal of cheminformatics期刊发表重要研究成果"claire: a contrastive learning-based predictor for ec number of chemical reactions"。在该研究成果中,团队利用对比学习,数据扩增,以及基于化学反应预训练模型的特征提取(embedding)策略,构建了一个用于预测ec分类编号的高效人工智能模型(claire)。

文章上线截图
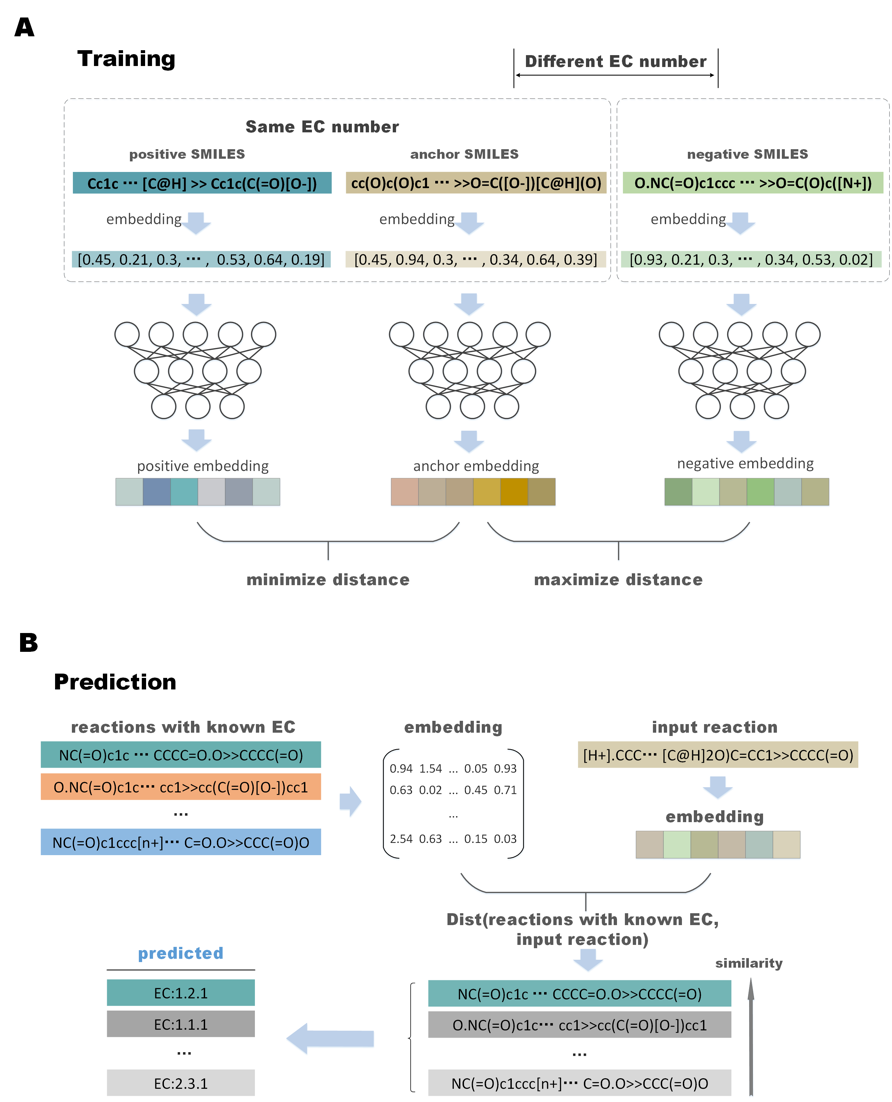
图1. claire的对比学习示意图
作者将claire与当前最领先的theia模型进行了对比。theia是2023年由瑞士洛桑联邦理工学院的科学家daniel probst发表在journal of cheminformatics期刊上的基于常规深度学习的模型——然而常规深度学习方法不能有效解决数据不平衡的问题。借助对比学习和数据扩增的策略,claire展现出了优异的性能——在测试集上,claire比theia有数倍的准确率提升,且在三级ec分类编号预测之间的一致性也显著高于theia。此外,作者利用酵母菌的代谢模型构建了另一个大型独立测试集。在该数据集中,claire的表现也显著高于theia。
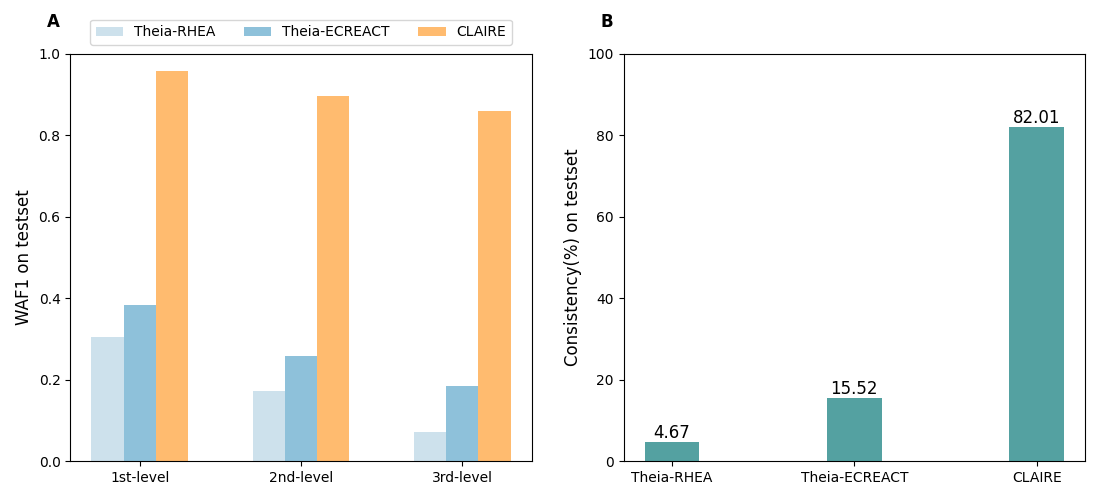
图2.claire在测试数据集上比theia表现更好
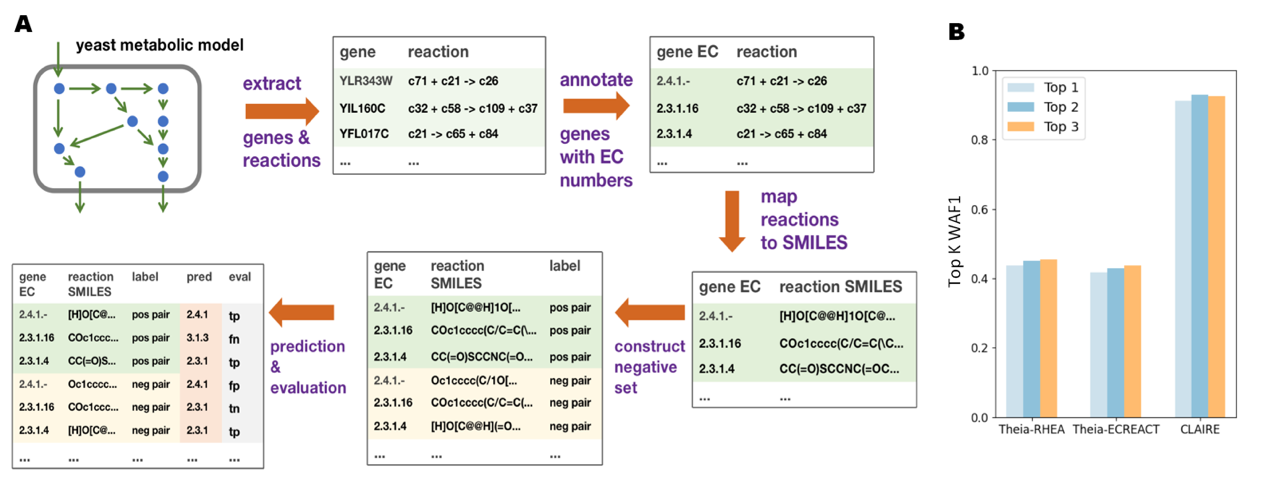
图3.claire在酵母菌代谢模型数据集上比theia表现更好
通过一系列严格的评估,研究人员展示了claire的强大能力:在酵母代谢模型中,它成功区分了真实的酶-反应配对与错误配对。代谢模型是生物体内代谢反应的定量化表示,涵盖基因、酶、代谢物及其细胞内分布,广泛应用于代谢工程和通量平衡分析等领域。claire的加入使得研究人员能够更高效地分析和注释反应网络,为代谢研究提供了全新可能。
此外,claire在逆合成路径规划和药物代谢预测等关键领域展示出巨大应用潜力。逆合成预测旨在推断生成目标化合物所需的原料及反应路径。在这一过程中,多个中间产物可能生成大量候选反应。通过claire预测的ec编号,可为这些反应分配相关酶,大幅提升最终目标化合物成功合成的可能性。另外,药物在人体内的代谢转化及路径是评估其安全性和有效性的重要环节。通过对潜在反应注释ec编号,claire能够清晰描绘可能的药物代谢路径,为毒性评估及药物开发提供有力支持。总而言之,该项成果在代谢工程和合成生物学领域中有着广泛的应用。
中国科学院深圳先进技术研究院合成生物学研究所研究员罗小舟为本文的通讯作者,团队成员博士后曾梓硕为文章的第一作者。该工作获得了深圳合成生物学创新研究院的支持。


